En el núcleo de cada coronavirus SARS-CoV-2 existe una molécula de ARN que trenza 30.000 nucleótidos. En esta secuencia se halla implícito el manual de instrucciones del virus. Las células humanas infectadas se ven obligadas a leer estas directrices genéticas y sintetizar 29 tipos de proteínas que se ensamblan para formar nuevas partículas víricas (viriones). Cuando se llena de partículas víricas, la célula muere y los virus infectan nuevas células siguiendo una progresión geométrica.
Sin embargo, cada vez que el ARN se copia, existe la posibilidad de que se produzcan errores (mutaciones) que, bien van en contra del propio virus, haciéndolo inviable, o bien le son favorables, al generarse nuevas versiones que escapan al sistema inmune del paciente durante algún tiempo o, lo que es peor, comprometen la eficacia de las vacunas. Las mutaciones dañinas para la viabilidad del virus tienden a desaparecer, mientras aquellas que favorecen su mantenimiento y propagación persistirán.
Un linaje del coronavirus SARS-CoV-2 (como cualquier otro virus) acumulará varias mutaciones en un breve período temporal, dada la rapidez de su multiplicación exponencial.
MUTACIÓN B.1.1.7
La variante notificada en Reino Unido acumula 17 mutaciones que sustituyen o eliminan determinados aminoácidos de las proteínas virales. Fue designado Variant of Concern 202012/01 por el Public Health England. Es parte del linaje B.1.1.7 del SARS-CoV-2.
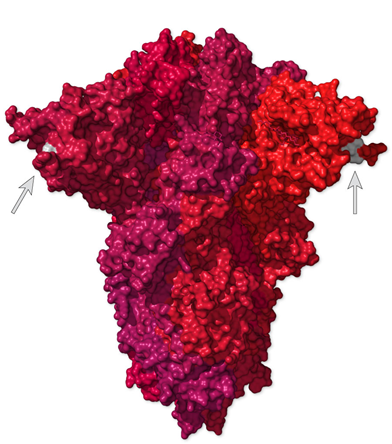
OCHO MUTACIONES QUE AFECTAN A LA PROTEÍNA S (SPIKE)
Estas son las mutaciones más preocupantes, ya que alteran la conformación (estructura tridimensional) de la proteína S de la superficie del virus, responsable de la infección.
La proteína S está formada por tres polipéptidos entrelazados que, en conjunto, totalizan 1.273 aminoácidos. Las proteínas S de la variante B.1.1.7 tienen dos deleciones y seis sustituciones en su secuencia de aminoácidos. Estos cambios (deleciones y sustituciones) modifican la estructura 3D de la proteína y la interacción con los receptores de las células humanas. [Los receptores de las células son ACE2, acrónimo de Angiotensin Convertase Enzyme type 2].
MUTACIÓN N501Y DE LA PROTEÍNA S
Se sospecha que la mutación N501Y es muy importante para que el SAS-CoV-2 que, de esta manera, incrementa la contagiosidad de la cepa B.1.1.7. La denominación (N501Y) se debe a la sustitución del aminoácido N (asparagina) en posición 501 por el aminoácido Y (tirosina) en dos de los tres polipéptidos que conforman la proteína S. Esta sustitución se produce en una posición clave de la proteína S para su interacción con los receptores de la membrana celular. La consecuencia es la mayor afinidad por los receptores ACE2 de la membrana celular.
Se considera que la mutación N501Y ha evolucionado de forma independiente en muchos linajes del SARS-CoV-2. Además del linaje B.1.1.7, la mutación se ha producido en otras cepas de Australia, Brasil, Dinamarca, Japón, Países Bajos, Sudáfrica, Gales, y algunos estados de Estados Unidos (Illinois, Texas, Luisiana y Ohio).
DELECIÓN H69-V70 DE LA PROTEÍNA S
Una misteriosa mutación en la cepa B.1.1.7 elimina los aminoácidos 69 y 70 de la proteína S. Esta doble deleción permite al SARS-CoV-2 infectar las células con mayor eficiencia, tal vez dificultando la unión de los anticuerpos. [H69 se refiere al aminoácido histidina en posición 69; y V70, al aminoácido valina en posición 70]. Estas mutaciones afectaron a visones en Dinamarca. [Dinamarca tiene la mayor crianza de estos animales en todo el mundo, anunciando que exterminaría a toda su población de visones, decisión que todavía no ha llevado a término].
DELECIÓN Y144/145 DE LA PROTEÍNA S
Esta deleción se refiere a los aminoácidos tirosina (Y) en las posiciones 144 o/y 145 de dos polipéptidos que conforman la proteína S. Se sabe muy poco, especulándose que puedan dificultar la unión de los anticuerpos.
MUTACIÓN P681H DE LA PROTEÍNA S
Estas mutaciones afectan a los aminoácidos P o H situados en el tronco de la proteína S (la región que se inserta en el esferoide vírico).
Cuando las proteínas S se ensamblan en el núcleo del virus, deben experimentar un corte en el tallo de la espiga (proteína S)
MUTACIÓN DE INTERRUPCIÓN ORF Q27 (STOP ORF Q27)
La proteína ORF8 es una de las más misteriosas del SARS-CoV-2. En un experimento se eliminó la proteína, pero el virus continuaba propagándose. Se infiere que la proteína ORF8 no es imprescindible para la replicación vírica, pero su eliminación podría de alguna manera ser ventajosa [para el virus].
ORF8 es una pequeña proteína, con solo 121 aminoácidos. La mutación elimina el aminoácido en posición 27, generándose una señal de stop génico. Los virus con esta mutación tienen un muñón de 26 aminoácidos en lugar de los 121 de la proteína completa.
DETECCIÓN Y PROPAGACIÓN
 La cepa mutante B.1.1.7 surgió (o se detectó) por primera vez en Reino Unido a finales de noviembre de 2020, si bien, tras analizar muestras anteriores, su presencia se retrotrae a 20 de septiembre.
La cepa mutante B.1.1.7 surgió (o se detectó) por primera vez en Reino Unido a finales de noviembre de 2020, si bien, tras analizar muestras anteriores, su presencia se retrotrae a 20 de septiembre.
Desde entonces esta mutación se ha detectado en más de 50 países. Se estima que la nueva cepa aumenta la contagiosidad en más del 50%, si bien no parece más virulenta.
¿CÓMO SURGIÓ Y EVOLUCIONARÁ LA VARIANTE B.1.1.7?
Se supone que la mutación se produjo en una única persona, tal vez alguien con algún tipo de inmunodeficiencia. Las personas con déficits inmunitarios albergan el virus durante largos períodos de tiempo (meses) permitiendo que el virus, bajo la presión de selección de los tratamientos o suero hiperinmune, experimente mutaciones.
OTRAS MUTACIONES
Una de las primeras mutaciones que inquietó a la comunidad internacional se designó D614G. Surgió en la República Popular China a comienzos de la pandemia, y pudo ser responsable de la rápida propagación del virus (compárese con la epidemia del SARS-CoV-1 durante los años 2002-2004). En muchos países la variante D614G fue la cepa principal. De hecho, la mutación británica (B.1.1.7) deriva de la cepa D614G.
Todavía en fechas más reciente otra mutación surgida en Sudáfrica conocida como 501Y.V2, se extendió muy rápidamente por varios países. La variante sudafricana es portadora de ocho mutaciones que cambian la estructura aminoacídica de la proteína S. Una de estas ocho mutaciones es la N501Y citada al comienzo del artículo.
Por ahora, todas estas mutaciones no eluden la acción de las vacunas. Sin embargo, mientras el virus permanezca en la comunidad se seguirán produciendo mutaciones (espontáneas o inducidas por los tratamientos y/o las vacunas). Es, pues, prioritario que el virus deje de circular en todo el mundo, o su prevalencia sea residual, antes de que las vacunas dejen de ser eficaces.
Zaragoza, a 19 de enero de 2021
Dr. José Manuel López Tricas
Farmacéutico especialista Farmacia Hospitalaria
Farmacia Las Fuentes
Zaragoza