Resultado de una electroforesis en gel de ADN tratado con colorante fluorescente. Dado que la carga negativa neta de la molécula es prácticamente idéntica, la migración diferencial en el gel soporte depende de su forma; y ésta es función del superenrollamiento, determinado por su «índice de enlace» (ver texto más adelante bajo el epígrafe «Aspectos topológicos del ADN».
Un estudio realizado en una familia afectada de sindactilia (fusión congénita de dedos de manos y/o pies) está permitiendo descubrir aspectos genéticos desconocidos hasta ahora, con trascendentes implicaciones en un creciente número de enfermedades.
Las mutaciones de esta familia afectada de sindactilia involucran a una característica de diseño del ADN que se ha dado en denominar como TAD, acrónimo en inglés de Topologically Associating Domains. La cadena de ADN se estructura conceptualmente en fragmentos de extensión variable que denominamos genes. Pero, el ADN también parece organizarse en «dominios topológicos» (TAD).
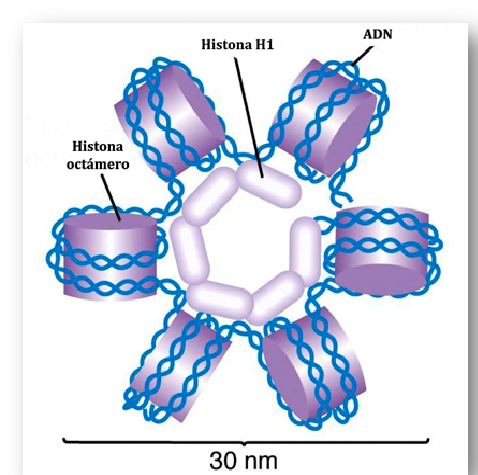
El concepto de TAD surge del conocimiento de la estructura tridimensional del genoma esto es, de su topología. El ADN humano es una cadena de tres mil millones de subunidades químicas repetitivas empaquetadas de modo exquisito dentro del núcleo celular. Si el ADN humano constreñido en el núcleo de cualquier célula pudiese extenderse mediría más de metro y medio. Empaquetado, su diámetro es de pocas mil millonésimas de metro. Para que el empaquetamiento sea posible, el ADN, una molécula con importante carga negativa neta, se asocia con proteínas con carga positiva, las histonas. [1nm (nanómetro) = 10-9 metros, esto es, una mil millonésima de metro].
El complejo «ADN-histonas» se denomina cromatina, por su reacción a la tinción con colorantes, denominación que surgió durante los primeros estudios microscópicos tiempo ha.
Como es bien conocido el ADN es una doble hélice formada por 2 cadenas anti-paralelas complementarias. Una magnitud importante es el «número de enlace» (Lk, de Linking) definido como el número de veces que una cadena rodea completamente a la otra. El «número de enlace» es una propiedad topológica, no geométrica. La principal característica de una propiedad topológica es su invariabilidad cuando solo cambia la forma de la estructura. Cuando la estructura se escinde y se vuelve a unir de una forma diferente, el «número de enlace» pude variar, pero a costa de otros dos parámetros topológicos, la «torsión» y el «giro» (véase más adelante en este mismo texto, bajo el epígrafe «Aspectos topológicos del ADN».
Los ahora denominados TAD (Topologically Associating Domains) se hallan en la base del protocolo de plegamiento del ADN. Desde el desciframiento de la estructura de la molécula de ADN y su vinculación con el código genético durante la década de 1950, esta molécula se ha considerado una especie de «código informático», en la que la secuencia de nucleótidos determina la secuencia de aminoácidos, mediante un proceso complejo y relativamente bien descifrado.
ASPECTOS TOPOLÓGICOS BÁSICOS DEL ADN
 ADN superenrollado de distintas formas (microscopio electrónico)
ADN superenrollado de distintas formas (microscopio electrónico)
La topología es una rama de las matemáticas que estudia propiedades estructurales invariantes por deformidad, sobre todo estiramientos o doblamientos.
La propiedad topológica fundamental de una molécula de ADN circular (como la de muchos bacteriófagos en donde se iniciaron los estudios) es su «número de enlace». Se designa Lk (de Linking); y equivale al número de veces que una hebra de ADN se enrolla en sentido dextrógiro alrededor del eje de la doble hélice cuando el eje se constriñe a un plano. En una molécula de ADN circular (sin solución de continuidad) el número de enrollamientos es 25 (Lk = 25). Para una molécula de ADN parcialmente desenrollada Lk = 23 (porque se ha desenrollado dos veces). Este mismo valor de Lk se aplica a una molécula superenrollada (ha tenido que desenrollarse dos veces antes de superenrollarse de nuevo). Se generan así «isómeros topológicos» (topoisómeros) que solo se diferencian en el número de enlaces. Los «isómeros topológicos» son inter-convertibles, pero para ello las hebras han de escindirse (1 o las dos hebras de la doble hélice del ADN) y volverse a unir en una conformación topológica diferente.
Los «isómeros topológicos» no lo son desde el punto de vista geométrico. Aun cuando tienen el mismo valor de Lk (número de enrollamientos), difieren en otros dos parámetros topológicos, el giro (Tw – del inglés Twist -) y la torsión (Wr – del inglés Writhe -). [El giro (Tw) es una medida del grado de enrollamiento helicoidal; y la torsión (Wr) es una medida del enrollamiento de la doble hélice]. Matemáticamente se puede demostrar que Lk = Tw + Wr. También se puede demostrar termodinámicamente que la «energía libre» alcanza un valor mínimo cuando el 70% aproximadamente de Lk se refleja en torsión (Wr); y el 30% aproximadamente en giro (Tw)]. La mayor parte de las moléculas de ADN presentes en la naturaleza están superenrolladas negativamente (enrolladas en sentido dextrógiro, girando de igual forma que un tornillo cuando se clava con un berbiquí). ¿Por qué? El superenrollamiento negativo facilita la apertura de la doble hélice para la replicación y transcripción. En cambio, el superenrollamiento positivo (levógiro) condensa la molécula de ADN tornándola inaccesible a las enzimas involucradas en la replicación y la transcripción.
La conversión entre «isómeros topológicos» es un proceso catalizado por enzimas, denominadas por esta razón topoisomerasas. Las topoisomerasas son moléculas biológicas que hacen matemáticas. Las primeras enzimas de esta clase fueron descubiertas por James Wang y Martin Gellert. Se trataba de las topoisomerasas tipo I. Éstas escinden una sola hebra de ADN. Por el contrario, las topoisomerasas tipo II dan lugar al clivaje de las dos hebras de ADN. Las topoisomerasas tipo II, también denominadas ADN-girasas, son la diana farmacológica de algunos medicamentos, tales como el ácido Nalidíxico y las quinolonas quimioterapéuticas (Ciprofloxacino y análogos). Estos medicamentos inhiben la ADN-girasa de procariotas (bacterias y hongos) de manera preferencial a la ADN-girasa de las células eucariotas. Su mecanismo antibacteriano radica en esta acción farmacodinámica. Camptotecina, un fármaco antineoplásico, inhibe la topoisomerasa I humana, mediante la formación de una unión covalente con el complejo enzima-ADN.
LEER ENTRE PLIEGUES
Hasta ahora la mayoría de las enfermedades genéticas se asocian con mutaciones en la secuencia de nucleótidos (sustitución o deleción de uno o varios nucleótidos). Los genes mutantes codifican proteínas erróneas que no pueden llevar a cabo la función o su grado de eficacia bioquímica es deficiente. Así, por ejemplo, la «Distrofia Muscular de Duchenne» está causada por la síntesis de una proteína muscular defectuosa. Las consecuencias son devastadoras para la persona afectada.
En la «enfermedad de Huntington» (antes denominada «Corea de Huntington») se presentan fragmentos repetidos de ciertos nucleótidos que mancillan el gen que codifica la síntesis de otra trascendente proteína cerebral. Estos oligonucleótidos adicionales resultan ser neurotóxicos. Y el resultado de esta alteración genética también tiene efectos dramáticos para las personas afectadas.
Es intelectualmente cómodo pensar que la información genética es lineal y unidireccional. Bajo este criterio simplista, las enfermedades genéticas se deben a una mutación que se trasunta en la síntesis de una proteína incorrecta y no funcional.
La secuenciación del genoma humano completo ha mostrado que para la síntesis de la mayoría de las proteínas importantes del organismo apenas se precisa el 3% de las tres mil millones de pares de bases de nucleótidos que estructuran el ADN humano. ¿Qué función tiene el llamado «ADN silente»?
Existe otra cuestión no resuelta: ¿por qué el ADN humano se organiza en 23 fragmentos (cromosomas) duplicados. Cada una de las dos copias de un cromosoma se denomina alelo.
Tal vez debemos considerar el genoma como una compacta «bola» de cromosomas, que asociado a las proteínas histonas, constituyen la cromatina. Pero esta «bola» compacta está en permanente actividad, desorganizándose de manera parcelada, transcribiéndose en ARN (el molde a partir del que se sintetizan las proteínas), y replegándose de nuevo.
 De cuando en cuando, la división celular (mitosis) da lugar a procesos de mayor calado: los cromosomas se separan y migran a los extremos de la célula («huso acromático», véase representación), se duplican en su integridad; y se reubican con precisión de tal suerte que cada célula hija recibe un número idéntico de cromosomas que la célula madre. ¡Increíblemente perfecto! En todos estos procesos no hemos de obviar a las histonas, esas proteínas que estabilizan eléctricamente la nucleofílica molécula de ADN. [El ADN es una molécula con importante carga negativa neta].
De cuando en cuando, la división celular (mitosis) da lugar a procesos de mayor calado: los cromosomas se separan y migran a los extremos de la célula («huso acromático», véase representación), se duplican en su integridad; y se reubican con precisión de tal suerte que cada célula hija recibe un número idéntico de cromosomas que la célula madre. ¡Increíblemente perfecto! En todos estos procesos no hemos de obviar a las histonas, esas proteínas que estabilizan eléctricamente la nucleofílica molécula de ADN. [El ADN es una molécula con importante carga negativa neta].
La genética quiere mirar más allá, indagando en la relación entre la conformación y la actividad de cada porción del genoma en un momento dado. Una cuestión inicial que hay que resolver es ¿cómo se pliega la doble hélice del ADN?; o dicho de otra manera: ¿cuáles son sus diferentes y complejas conformaciones espaciales? Para este estudio es preciso recurrir a la rama de las matemáticas denominada topología.
Un equipo de investigación dirigido por el Dr. Dekker ha diseñado la técnica de «captura de conformación cromosómica». Esta tecnología permite el seguimiento de la estructura profunda del ADN. Para ello se “congela” la estructura de la cromatina en un momento dado (“congelar” en el sentido de bloquear su permanente actividad como si se tratase de una imagen fija). A continuación se disecciona enzimáticamente y se “descongela” la cromatina para que retome su función.
Mediante estos estudios de «conformación cromosómica» se ha desentrañado algo más de la organización genómica. Cada cromosoma está estructurado en alrededor de dos mil TAD (Topologically Associating Domains) de muy distintos tamaños. De alguna manera los TAD contienen los códigos para el plegamiento de esta gran molécula.
Los mecanismos de control génico son complejos y poco conocidos. Aun descubriendo los patrones generales, seguiríamos ignorando la función de una gran parte del genoma.
Hasta donde creemos saber los TAD parecen gozar de cierta autonomía dentro de la estructura completa de la doble hélice del ADN. Los genes «controladores», «reguladores» e «inhibidores» están próximos a los genes «codificadores» (los que se trascriben y traducen en proteínas).
TRASPASANDO FRONTERAS
La compartimentación del ADN en estos TAD previene de alguna manera la comunicación errónea entre genes. Para ello (o gracias a ello) determinadas proteínas denominadas genéricamente «cohesinas» y CTCF (acrónimo en inglés de «CCCTC.binding Factor») separan y aíslan las TAD a la manera de bolardos genéticos. Estas proteínas contribuirían a que se produzcan «enredos» en el extremadamente complejo plegamiento de la molécula de ADN.
La mejor evidencia de la importancia de los TAD («Topologically Associating Domains») es observar qué sucede cuando se desestructuran. La pérdida de los límites de estos «dominios genéticos» está asociada con determinados tipos de deriva cancerosa en órganos tan diversos como el colon, esófago, cerebro y sangre. En estos tumores no se han hallado las mutaciones habituales asociadas a las de genes codificadores de proteínas específicas. Las mutaciones se observan en los genes que delimitan los TAD en el conjunto de la molécula de ADN.
Un ejemplo de este tipo de tumores es descrito en un reciente trabajo publicado en la revista Science. Se trata de una mutación en el locus al que se adhieren las proteínas que antes hemos denominado «cohesinas». Este locus se halla próximo a un gen designado como TAL1 (acrónimo de: «T-cell Acute Lymphocitc leukemia protein-1»). Se sabe que la activación extemporánea de este gen (proto-oncogén) causa leucemia.
Conforme mayor conocimiento se adquiera de los TAD, probablemente se hallará explicación de muchas malformaciones, como la sindactilia; así como de un sinnúmero de neoplasias de origen hoy día desconocido.
Edith Heard, directora del Departamento de Genética y Biología del Desarrollo del Instituto Curie (Francia) quien, junto al Dr. Jared Dekker acuñó el concepto TAD (Topologically Associating Domains) afirmó que se trata de profundizar en la comprensión de la estructura del ADN, soporte químico de un constructo mucho más complejo, el genoma.
Zaragoza, 20 de enero de 2017
Dr. José Manuel López Tricas
Farmacéutico especialista Farmacia Hospitalaria
FARMACIA LAS FUENTES
ZARAGOZA